Metodos de secuenciación de ADN
Secuenciar una molécula de ADN consiste en determinar en qué orden se disponen los cuatro nucleótidos (A, T, C y G) que componen la molécula. A partir de 1977 dos técnicas han sido utilizadas habitualmente para la secuenciación de moléculas largas de ADN, por una parte el método de Alan Maxan y Walter Gilbert de hidrólisis química especifica y por otra la de interrupción controlada con didesoxinucleótidos de Frederic Sanger.
El método de degradación química (Maxam y Gilbert, 1977)
En este método, un fragmento de ADN de cadena doble o sencilla se marca en los extremos 5´ o 3´ de una o ambas hebras con 32P. Después, la muestra de ADN se divide en cuatro alícuotas y se fragmenta en cuatro reacciones químicas distintas. Posteriormente, los fragmentos de ADN generados pueden ser separados por electroforesis en cuatro carriles distintos con base en su tamaño. Conociendo el nucleótido en el que se realizaron los cortes, se puede inferir la secuencia de la molécula original. Las reacciones químicas que se utilizan para fragmentar la molécula de ADN son las siguientes:
1. Corte de las purinas. Las purinas adenina y guanina se metilan con dimetil sulfato (DMS). Después, la reacción es tratada en condiciones alcalinas; la molécula de ADN se fragmenta en las purinas metiladas. Como resultado, se obtiene una serie de bandas oscuras que corresponden a las guaninas (las cuales se metilan 5 veces más rápido), y bandas claras que corresponden a las adeninas. Para interpretar fácilmente el patrón de bandasgeneradas, se puede comparar contra un tratamiento que favorezca el corte de las adeninas.
2. Corte de adeninas. Esta reacción es una variación de la anterior. Las purinas metiladas se tratan inicialmente con un ácido diluido. Esto favorece el corte de las adeninas metiladas. Después de un tratamiento alcalino las guaninas también son cortadas. Este tratamiento genera una serie de bandas oscuras y claras que también corresponden a las adeninas, y las guaninas, respectivamente.
3. Corte de pirimidinas. Esta reacción utiliza el reactivo hidracina, que corta las bases citosina y timina. Posteriormente, se trata con piperidina para completar la reacción.
4. Corte de citosina. La presencia de NaCl 2M inhibe la reacción de hidracina con tiamina, y el tratamiento posterior con piperidina, produce solamente fragmentos que terminan en citosina.
El método enzimático (Sanger et al., 1977)
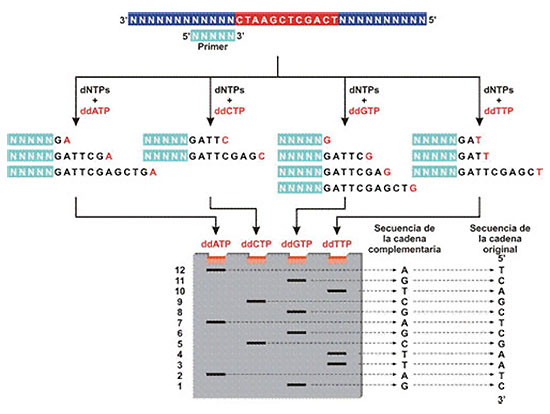
La técnica enzimática se basa en la interrupción controlada de la replicación del ADN in vitro. Se realiza una PCR del fragmento que se va a secuenciar pero, a diferencia de una PCR convencional, se utiliza un solo iniciador y se agregan dideoxinucleótidos (ddNTPs) que carecen del grupo hidroxilo en el extremo 3´ y están marcados con radiactividad o con fluoróforos. Cuando se incorpora un dideoxinucleótido a la cadena en elongación, se termina su amplificación, de tal manera que al final, se tienen fragmentos de diferente longitud, cada uno terminando en un ddNTPs. Los fragmentos obtenidos, se separan y analizan electroforéticamente de manera manual o automática.
En el método de Sanger (1977), la estrategia es hacer cuatro reacciones diferentes de síntesis de ADN, utilizando un ddNTP distinto en cada tubo. Con la mezcla del nucleótido normal (dNTP) y su 21 terminador (ddNTP), se pueden generar fragmentos complementarios de diferentes tamaños que terminan en el mismo nucleótido. Después, estos fragmentos se pueden separar en un gel de electroforesis con cuatro carriles distintos, para determinar la secuencia del templado.
Referencias:
Fundamentos de bioquímica estructural. 2006. Teijón Rivera, J.M. Editorial Tebar. 444 pp



Comentarios
Publicar un comentario